TIANSeq Fast DNA Library Kit (ілюмінатар)


Асаблівасці
- Добрая аднастайнасць паслядоўнасці: Няма зрушэння асновы працэсу фрагментацыі ДНК і працэсу ампліфікацыі ПЦР.
- Высокая эфектыўнасць пераўтварэння бібліятэкі: Высокаэфектыўная канструкцыя бібліятэкі можа быць забяспечана толькі за 0,25 нг узораў ДНК.
- Хуткая праца: увесь працэс падрыхтоўкі бібліятэкі займае ўсяго 2 гадзіны.
Спецыфікацыя
Тып: Падрыхтоўка бібліятэкі ДНК да платформы высокапрадукцыйнага секвенирования illumina.
Узор: кдДНК або невялікія фрагменты ДНК пасля стрыжкі праз ультрагукавую або ферментатыўную апрацоўку.
Мэта: Двухцепочечная ДНК.
Пачатковы ўзор уводу: 0,25 нг- 1 мкг.
Час працы: 2-2,5 гадзіны.
Ніжэйшыя праграмы: Размяшчэнне на платформе illumina.
s
Усе прадукты можна наладзіць для ODM/OEM. Для падрабязнасцей,калі ласка, націсніце Індывідуальнае абслугоўванне (ODM/OEM)

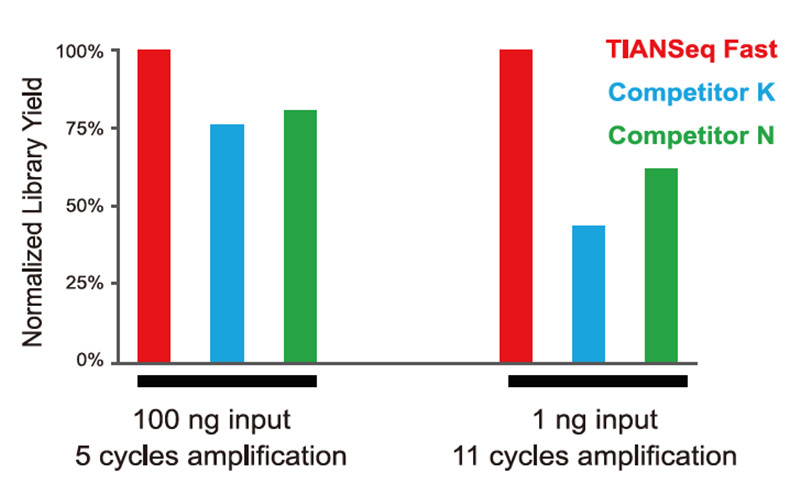 |
Параўнанне ўраджайнасці будаўніцтва бібліятэк Малюнак 1. Параўнанне ўраджайнасці бібліятэк сярод розных прадуктаў |
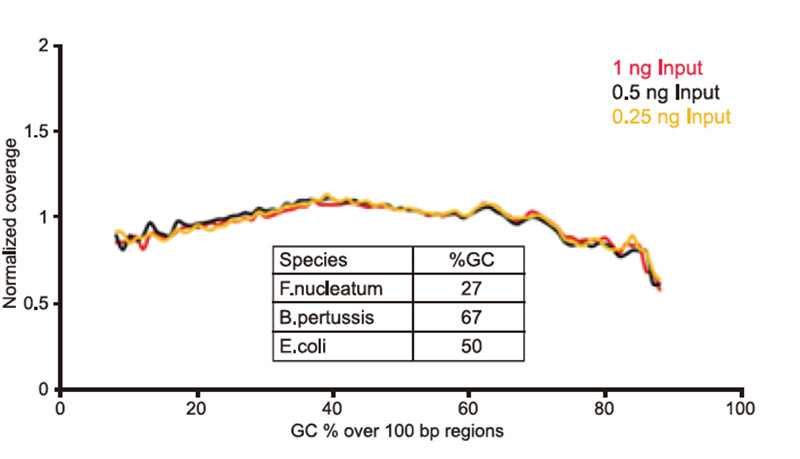 |
Ахоп геному нізка ўваходнай ДНК Малюнак 2. Вынікі ахопу геному нізкай ДНК. Параўнанне паказчыкаў ахопу розных уваходных ДНК (1,0,5,0,25 нг) трох сумесяў бактэрыяльнага геному з розным утрыманнем ГХ у тым жа малярным суадносінах. Вынікі паказваюць, што набор хуткай бібліятэкі ДНК TIANSeq гарантуе добрую стабільнасць і высокі каэфіцыент канверсіі для пабудовы бібліятэкі ўзораў слядоў. |
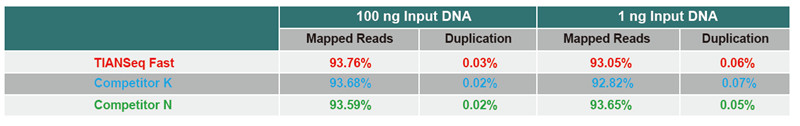 |
Параўнанне дадзеных паслядоўнасці розных прадуктаў |
У цяперашні час высокапрадукцыйная тэхналогія секвенирования ў асноўным заснавана на тэхналогіі секвенирования наступнага пакалення. Паколькі працягласць чытання тэхналогіі паслядоўнасці наступнага пакалення абмежавана, мы павінны разбіць поўную даўжыню на невялікія бібліятэкі фрагментаў. У адпаведнасці з патрэбамі розных эксперыментаў паслядоўнасці, мы звычайна выбіраем адзінкавае або двухканцовае паслядоўнасць. У цяперашні час фрагменты ДНК бібліятэкі секвенування наступнага пакалення звычайна распаўсюджваюцца ў дыяпазоне 200-800 bp.

а) ДНК дрэннай якасці і змяшчае інгібітары. Выкарыстоўвайце якасныя ўзоры ДНК, каб пазбегнуць інгібіравання актыўнасці ферментаў.
б) Колькасць узору ДНК недастатковая пры выкарыстанні метаду без ПЦР для пабудовы бібліятэкі ДНК. Калі ўвод фрагментаванай ДНК перавышае 50 нг, працоўны працэс без ПЦР можа быць выбарачна выкананы ў працэсе стварэння бібліятэкі. Калі нумар копіі бібліятэкі занадта нізкі, каб яе можна было непасрэдна секвенировать, бібліятэку ДНК можна ампліфікаваць з дапамогай ПЦР пасля перавязкі адаптара.
в) забруджванне РНК прыводзіць да недакладнай пачатковай колькаснай ацэнкі ДНК. Заражэнне РНК можа існаваць у працэсе ачысткі геномнай ДНК, што можа прывесці да недакладнай колькаснай ацэнкі ДНК і недастатковай загрузкі ДНК падчас пабудовы бібліятэкі. РНК можна выдаліць, апрацаваўшы яе РНКазай.
А-1
а) З'яўляюцца невялікія фрагменты (60 б.п.-120 д.а.). Ачыстка з дапамогай магнітных пацерак Agencourt AMPure XP дазваляе эфектыўна выдаліць гэтыя фрагменты адаптара і забяспечыць якасць паслядоўнасці.
б) Вялікія фрагменты з'яўляюцца ў бібліятэцы пасля ПЦР -ампліфікацыі. Памер фрагмента ДНК бібліятэкі павялічыцца на 120 б.п. пасля перавязкі адаптара. Калі фрагмент ДНК павялічваецца больш чым на 120 б.п. пасля перавязкі адаптара, гэта можа быць выклікана анамальнай ампліфікацыяй фрагментаў пры празмернай ампліфікацыі ПЦР. Памяншэнне колькасці ПЦР -цыклаў можа прадухіліць сітуацыю.
в) Анамальны памер бібліятэчных фрагментаў ДНК пасля перавязкі адаптара Даўжыня адаптара ў гэтым наборы складае 60 пар. Калі два канцы фрагмента перавязуць на перахаднікі, даўжыня павялічыцца толькі на 120 б.п. Калі вы выкарыстоўваеце адаптар, які не прадугледжаны гэтым камплектам, звярніцеся да пастаўшчыка, каб даць адпаведную інфармацыю, напрыклад, даўжыню адаптара. Калі ласка, пераканайцеся, што працоўны працэс і эксплуатацыя эксперыменту выконваюць крокі, апісаныя ў кіраўніцтве.
d) Анамальны памер фрагмента ДНК да перавязкі адаптара Прычынай гэтай праблемы могуць быць няправільныя ўмовы рэакцыі падчас фрагментацыі ДНК. Для рознага ўводу ДНК варта выкарыстоўваць розныя тэрміны рэакцыі. Калі ўвод ДНК складае больш за 10 нг, мы рэкамендуем выбраць час рэакцыі 12 хвілін у якасці пачатку для аптымізацыі, а памер фрагмента, атрыманы ў гэты час, знаходзіцца ў асноўным у дыяпазоне 300-500 у.а. Карыстальнікі могуць павялічваць або памяншаць даўжыню фрагментаў ДНК на 2-4 хвіліны ў адпаведнасці з іх уласнымі патрабаваннямі, каб аптымізаваць фрагменты ДНК з неабходным памерам.
А-2
а) Час фрагментацыі не аптымізаваны Калі фрагментаваная ДНК занадта малая або занадта вялікая, калі ласка, звярніцеся да Кіраўніцтва па выбары часу фрагментацыі, прыведзенага ў інструкцыі для вызначэння часу рэакцыі, і выкарыстоўвайце гэты час у якасці кантролю, дадаткова наладзьце рэакцыйная сістэма, каб падоўжыць або скараціць 3 хвіліны, каб зрабіць больш дакладную карэкціроўку часу фрагментацыі.
А-3
Анамальнае размеркаванне ДНК па памеры пасля апрацоўкі фрагментацыяй
а) Няправільны метад размарожвання рэагента для фрагментацыі або рэагент не цалкам змешваецца пасля размарожвання. Размарозіць рэагент 5x Фрагментацыйная сумесь на лёдзе. Пасля размарожвання раўнамерна змяшайце рэагент, акуратна пстрыкнуўшы дном прабіркі. Не вірце рэагент!
б) Уваходная проба ДНК змяшчае ЭДТА або іншыя забруджвальныя рэчывы. Высіленне іёнаў солі і хелатирующих агентаў на этапе ачысткі ДНК асабліва важна для поспеху эксперыменту. Калі ДНК раствараецца ў 1 × TE, выкарыстоўвайце метад, прадстаўлены ў інструкцыі, для правядзення фрагментацыі. Калі канцэнтрацыя ЭДТА ў растворы нявызначаная, рэкамендуецца ачысціць ДНК і растварыць яе ў деионизированной вадзе для наступнай рэакцыі.
в) недакладнае пачатковае колькаснае вызначэнне ДНК Памер фрагментаванай ДНК цесна звязаны з колькасцю паступае ДНК. Перад апрацоўкай фрагментацыі неабходна дакладнае колькаснае вызначэнне ДНК з дапамогай Qubit, Picogreen і іншых метадаў, каб вызначыць дакладную колькасць ДНК у рэакцыйнай сістэме.
d) Падрыхтоўка рэакцыйнай сістэмы не адпавядае інструкцыі Падрыхтоўка раздробленай рэакцыйнай сістэмы павінна праводзіцца на лёдзе строга ў адпаведнасці з інструкцыямі. Для забеспячэння найлепшага эфекту ўсе кампаненты рэакцыі павінны быць змешчаны на лёд, а падрыхтоўка рэакцыйнай сістэмы павінна праводзіцца пасля поўнага астывання. Пасля таго, як падрыхтоўка завершана, пстрыкніце або піпеткай, каб старанна перамяшаць. Не вірце!
1. Няправільны метад змешвання (віхура, моцныя ваганні і г.д.) прывядзе да анамальнага размеркавання фрагментаў бібліятэкі (як паказана на наступным малюнку), што паўплывае на якасць бібліятэкі. Таму, калі рыхтуеце рэакцыйны раствор Fragmentation Mix, калі ласка, акуратна піпеткай уверх і ўніз змяшайце, або выкарыстоўвайце кончык пальца, каб пстрыкнуць і раўнамерна змяшаць. Будзьце асцярожныя, каб не змяшаць з віхром.

2. Для пабудовы бібліятэк неабходна выкарыстоўваць ДНК высокай чысціні
■ Добрая цэласнасць ДНК: дыяпазон электрафарэзу складае больш за 30 кб, без хвастоў
■ OD260/230:> 1.5
■ OD260/280: 1,7-1,9
3. Колькасць уваходнай ДНК павінна быць дакладнай. Для колькаснай ацэнкі ДНК прапануецца выкарыстоўваць метады Qubit і PicoGreen, а не Nanodrop.
4. Трэба вызначыць змест ЭДТА ў растворы ДНК. ЭДТА аказвае вялікі ўплыў на рэакцыю фрагментацыі. Калі ўтрыманне ЭДТА высокае, перад наступным тэстам неабходна правесці ачыстку ДНК.
5. Раствор для рэакцыі фрагментацыі неабходна прыгатаваць на лёдзе. Працэс фрагментацыі адчувальны да тэмпературы і часу рэакцыі (асабліва пасля дадання ўзмацняльніка). Каб забяспечыць дакладнасць часу рэакцыі, падрыхтуйце рэакцыйную сістэму на лёдзе.
6. Час рэакцыі фрагментацыі павінен быць дакладным Час рэакцыі на этапе фрагментацыі непасрэдна паўплывае на памер прадуктаў фрагментаў, уплываючы такім чынам на размеркаванне фрагментаў ДНК у бібліятэцы.
1. Які тып узору дастасавальны да гэтага набору?
Дастасавальны тып узору гэтага набору можа быць агульнай РНК або вычышчанай мРНК з добрай цэласнасцю РНК. Калі для стварэння бібліятэкі выкарыстоўваецца агульная РНК, рэкамендуецца спачатку выдаліць рРНК.
2. Ці можна ўзоры FFPE выкарыстоўваць для стварэння бібліятэкі з дапамогай гэтага набору?
МРНК ва ўзорах FFPE будзе дэградаваць у пэўнай ступені з адносна дрэннай цэласнасцю. Пры выкарыстанні гэтага набору для стварэння бібліятэкі рэкамендуецца аптымізаваць час фрагментацыі (скараціць час фрагментацыі або не выконваць фрагментацыю).
3. Выкарыстоўваючы крок выбару памеру, указаны ў інструкцыі да прадукту, што можа прывесці да ўстаўкі сегмента з невялікім адхіленнем?
Выбар памеру павінен ажыццяўляцца ў строгай адпаведнасці з крокам выбару памеру ў дадзеным кіраўніцтве па вырабе. Калі ёсць адхіленне, прычына можа заключацца ў тым, што магнітныя шарыкі не збалансаваны да пакаёвай тэмпературы ці не змешаны цалкам, піпетка недакладная або вадкасць засталася ў наканечніку. Для эксперыменту рэкамендуецца выкарыстоўваць наканечнікі з нізкай адсорбцыяй.
4. Выбар адаптараў у будаўніцтве бібліятэк
У камплекце для стварэння бібліятэкі няма рэагента-адаптара, і рэкамендуецца выкарыстоўваць яго разам з адаптарам TIANSeq з адным індэксам (Illumina) (4992641/4992642/4992378).
5. КК бібліятэкі
Колькаснае выяўленне бібліятэкі: Qubit і qPCR выкарыстоўваюцца для вызначэння масавай канцэнтрацыі і малярнай канцэнтрацыі бібліятэкі адпаведна. Аперацыя строга адпавядае інструкцыі да прадукту. Канцэнтрацыя бібліятэкі ў цэлым будзе адпавядаць патрабаванням паслядоўнасці NGS. Выяўленне дыяпазону размеркавання бібліятэк: Выкарыстоўвайце біяаналізатар Agilent 2100 для выяўлення дыяпазону распаўсюджвання бібліятэк.
6. Выбар нумара цыклу ўзмацнення
Згодна з інструкцыямі, колькасць ПЦР-цыклаў складае 6-12, а колькасць неабходных ПЦР-цыклаў трэба падбіраць у адпаведнасці з уводам пробы. У бібліятэках з высокім ураджаем пераўзмацненне звычайна адбываецца ў рознай ступені, што выяўляецца крыху большым пікам пасля піку мэтавага дыяпазону пры выяўленні біяаналізатара Agilent 2100 або выяўленай канцэнтрацыі Qubit ніжэйшай, чым у qPCR. Мяккае ўзмацненне - нармальная з'ява, якое не ўплывае на паслядоўнасць бібліятэк і наступны аналіз дадзеных.
7. Шыпы з'яўляюцца ў профілі выяўлення біяаналізатара Agilent 2100
З'яўленне ўсплёскаў пры выяўленні біяаналізатара Agilent 2100 абумоўлена нераўнамернай фрагментацыяй узораў, дзе будзе больш фрагментаў пэўнага памеру, і гэта стане больш відавочным пасля ўзбагачэння ПЦР. У гэтым выпадку прапануецца не праводзіць выбар памеру, гэта значыць усталяваць умову фрагментацыі на 94 ° C на працягу 15 хвілін інкубацыі, дзе размеркаванне фрагментаў невялікае і канцэнтраванае, і аднастайнасць можна палепшыць.
Катэгорыі прадуктаў
Чаму выбіраюць нас
З моманту свайго заснавання наша фабрыка распрацоўвае прадукты першага сусветнага класа з захаваннем прынцыпу
якасць у першую чаргу. Наша прадукцыя заваявала выдатную рэпутацыю ў прамысловасці і каштоўную давер сярод новых і старых кліентаў.










